Para dejar un comentario Regístrate aquí o si ya tienes una cuenta debes Iniciar sesión

Aves
agosto 10, 2020
Virus de Influenza, mucho más que solo Hemaglutinina
Lesly Romero Beltrán
Al escuchar hablar de los virus de influenza, pensamos inmediatamente en la Hemaglutinina (HA), y en cómo su sitio de corte puede determinar si los virus de influenza aviar pueden ser de baja o alta patogenicidad; pero los virus de influenza son partículas mucho más complejas, que requieren de la interacción de sus proteinas y genoma con gran parte del sistema de replicación de las células que infectan. Los virus influenza pertenecen a la familia Orthomyxoviridae. Los virus influenza A tienen un genoma de ácido ribonucleico (RNA) de cadena sencilla, en sentido negativo, de 8 segmentos. Algunos segmentos del genoma de influenza codifican para más de una proteína dependiendo del marco de lectura (1). Existen tres tipos de virus influenza A, B y C. La clasificación se basa en la secuencia de aminoácidos de la nucleoproteína (NP) y de la proteína de matriz 1 (M1). Los virus de tipo B y C son exclusivamente de humanos. Los virus tipo A puede infectar a humanos, otros mamíferos y aves, los principales reservorios de este tipo de virus son las aves migratorias (2). La clasificación de los subtipos de los virus de influenza A, depende de la combinación de dos proteínas de superficie, la HA y la Neuraminidasa (NA), la diferencia de nucleótidos entre los diferentes subtipos debe ser al menos un 30% (3).
El virus influenza A es pleomórfico, su diámetro varía de 50 a 120 nm (4), cuenta con una bicapa lipídica tomada de la membrana celular. En el interior del virión (Figura 1) se encuentra la proteína matriz 1, y la nucleocápside helicoidal; el genoma se encuentra empaquetado en estructuras conocidas como ribonucleoproteínas (RNP) conformadas por el RNA viral, la NP y el complejo de las polimerasas: Polimerasa básica 1 (PB1), polimerasa básica 2 (PB2) y polimerasa ácida (PA) (5). En el exterior, insertadas en la membrana lipídica, se encuentran las glicoproteínas HA y NA, que son los principales antígenos de superficie, y las proteínas virales que presentan la mayor diversidad aminoacídica en sus secuencias (6). La proteína de matriz 2 (M2) se encuentra en la membrana viral formando un canal iónico que permite la acidificación del virión y la liberación de las RNPs en el citoplasma de la célula infectada.
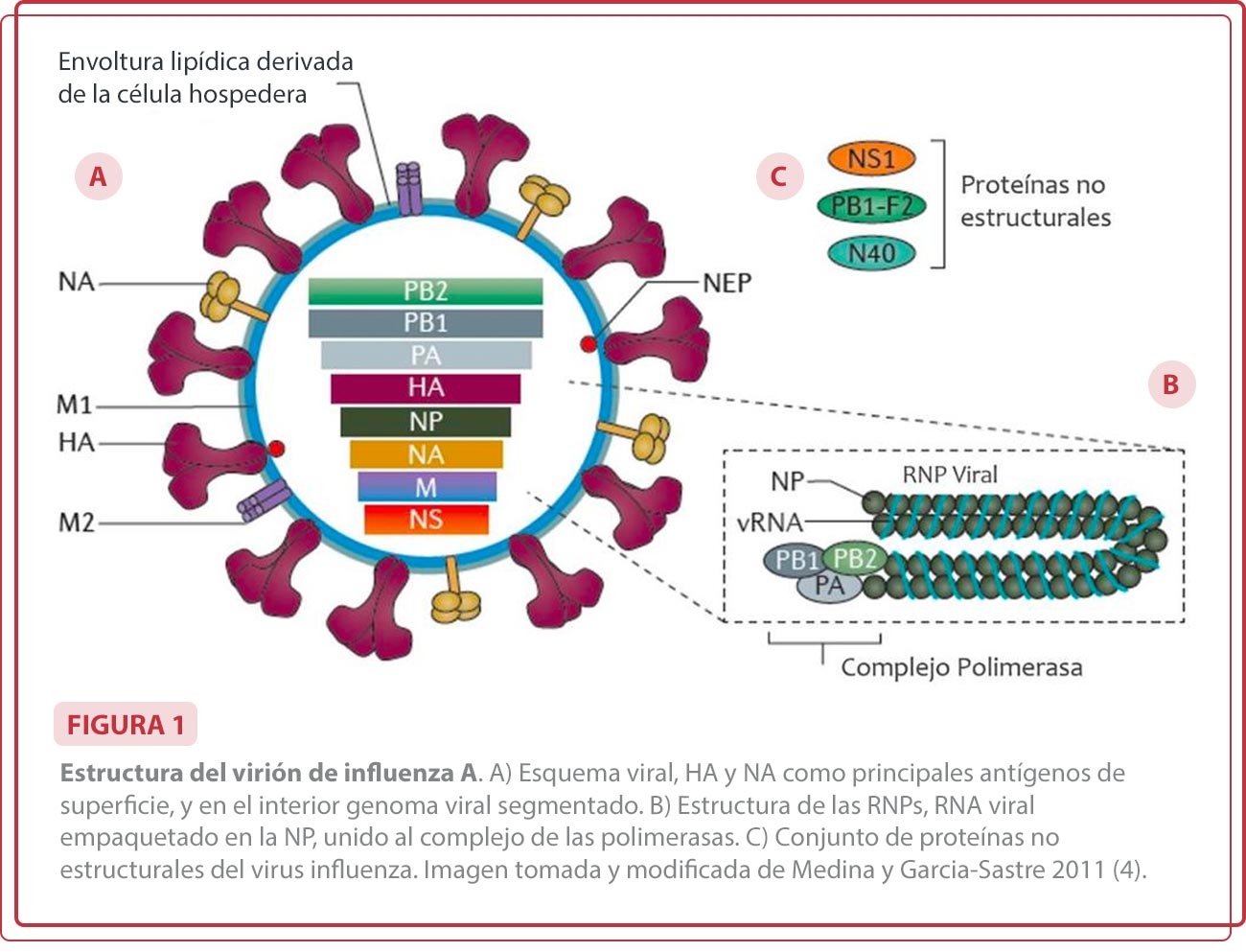
Proteínas virales
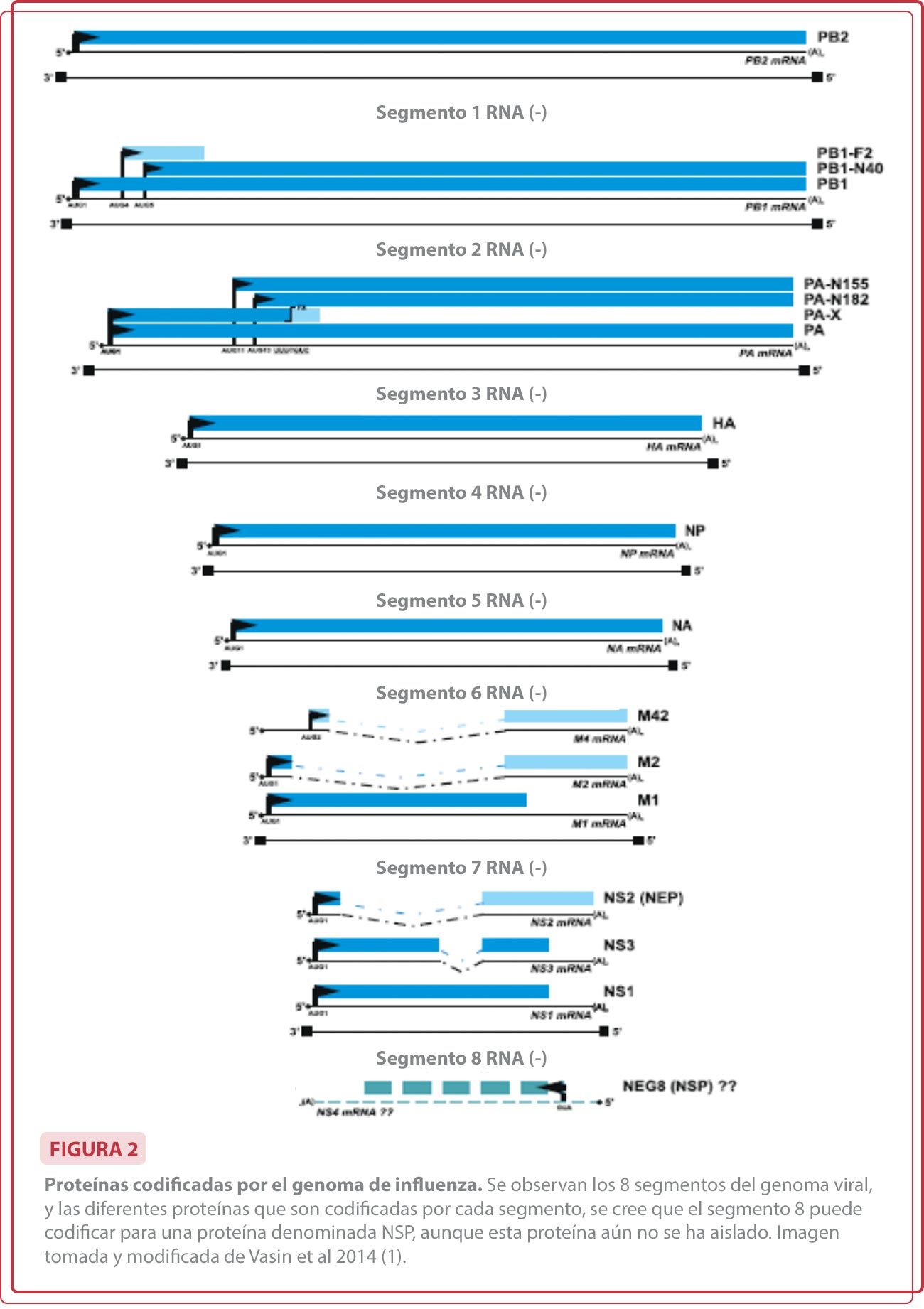
Las proteínas virales son codificadas por los ocho segmentos del genoma viral (1). Algunos segmentos codifican para más de una proteína, en esos casos se utilizan diferentes marcos de lectura para la transcripción de cada proteína (Fig. 2). La proteína PB2 es codificada por el segmento 1, forma parte del complejo de las polimerasas, está compuesta por 759 aminoácidos (aa), y su principal función es la transcripción del ácido ribonucleico viral (RNAv) a ácido ribonucleico mensajero (RNAm); reconoce a la polimerasa II celular (Pol II) y se une a ella, para secuestrar los RNAm celulares. También participa en la obtención de cap, que es una 7-metilguanosina en el extremo 5´ de los RNAm celulares, para su utilización en los RNAm virales (7).
Las proteínas que pueden ser codificadas por el segmento 2 son PB1, PB1 N40 y PB1-F2. PB1 forma parte del complejo de las polimerasas, está compuesta por 757 aa, y es la encargada de la elongación del RNAm y RNAv. PB1 N40, es una proteína no estructural de 718 aa, cuya función no ha sido bien determinada, se asocia con el balance en la expresión de PB1 y PB1-F2 (13), se considera un posible factor de virulencia (8). PB1-F2, es una proteína no estructural y proapoptótica de 90 aa, se presenta en algunas cepas de virus influenza, se asocia con incremento de virulencia, regulación de la replicación viral, favorecimiento de co-infección bacteriana, así como de modulación de la respuesta inmune (9).
Las proteínas que pueden ser codificadas por el segmento 3 son PA, PA-X, PA-N155 y PA-N188. La proteína PA compuesta por 716 aa forma parte del complejo de las polimerasas, tiene actividad endonucleasa y se encarga del corte de los RNAm del huésped para liberar cap, que se une al RNA transcrito del virus que se encuentra en sentido positivo, para ser reconocido por los ribosomas y actuar como RNAm. A ese proceso se le conoce como cap snatching (10). PA-X es una proteína compuesta por 232 aa, su función es reducir la acumulación de RNAm tanto del huésped como del virus debido a su actividad endonucleasa (11), así como inhibir la producción de proteínas celulares, utilizando la maquinaria de la célula para producir principalmente proteínas virales (12). Adicionalmente PA-X se ha asociado a la modulación de la respuesta inmune. Las proteínas PA-N155 compuesta por 568 aa y PA-N188 compuesta por 535 aa, no muestran actividad polimerasa, y aún se desconocen sus funciones, aunque se cree que PA-N155 juega un papel importante en el ciclo viral (1).
La NP es codificada por el segmento 5, está compuesta por 498 aa, su principal función es la de encapsular el RNAv, alrededor de 20 nucleótidos se unen a una subunidad de NP; las RNPs están conformadas por RNAv, empaquetado en NP, y unido a las polimerasas PB1, PB2 y PA (13). NP se une a M1 para el empaquetamiento de las RNPs en el virión. NP tiene función de helicasa (14), es el principal antígeno involucrado en la activación de los linfocitos T citotóxicos (15), y también interactúa con importinas, filamentos de actina, receptores de la CRM1 por sus siglas en inglés (Chromosome region maintenance 1).
Las proteínas M1, M2 y M42 son codificadas por el segmento 7. M1 compuesta por 252 aa, es el principal componente estructural de los viriones. Forma una capa intermedia entre la envoltura viral y las RNPs; Al asociarse a NEP participa en el transporte del genoma por el citoplasma, adicionalmente M1 dirige la gemación (16). M2 está compuesta por 97 aa, es una proteína integral de membrana, se encuentra glicosilada. Su principal función es la de canal iónico, permite el paso de protones al interior del virión durante la acidificación del endosoma celular, y de esta manera contribuye a los cambios conformacionales en HA, que desencadenan en el estrangulamiento de las membranas virales y endosomales y la liberación de los RNPs en la célula infectada (17). M42 está compuesta por 99 aa y es una isoforma de M2 con un ectodominio alternativo, su función es la misma, por lo que puede presentarse en combinación con M2, aunque se cree que M42 constituye solo un 0.2% de las proteínas virales (1).
La proteína No estructural 1 (NS1), proteína de exportación nuclear (NEP), proteína no estructural 3 (NS3) y posiblemente NSP, son codificadas por el segmento 8. NS1 es una proteína no estructural compuesta por 217 aa, su función es inhibir el Interferón que regula los mecanismos antivirales del huésped. NEP es un componente estructural de los viriones que se asocia a M1, está compuesta por 121 aa, y es la encargada de la exportación de las RNPs del núcleo celular, a través de la interacción con CRM1. NS3 es una isoforma de NS1compuesta por 174 aa, debido a una deleción de 43 aa; se asocia con adaptación en ratones (1). NSP es una proteína aún no aislada, que se cree puede estar compuesta de 156 a 216 aa, se considera que puede tener un papel importante en el ciclo viral, ya que constituye una región altamente conservada de 167 codones a partir de un origen de replicación denominado NEG8. Se ha descrito que los virus humanos que surgieron en 1948 extendieron la longitud de la secuencia codificante a 216 codones. Mediante análisis de predicción de la estructura proteica, se determinaron dos dominios transmembranales, múltiples sitios de fosforilación, un sitio de glicosilación, un motivo de cremallera de leucina, y un péptido señal (1).
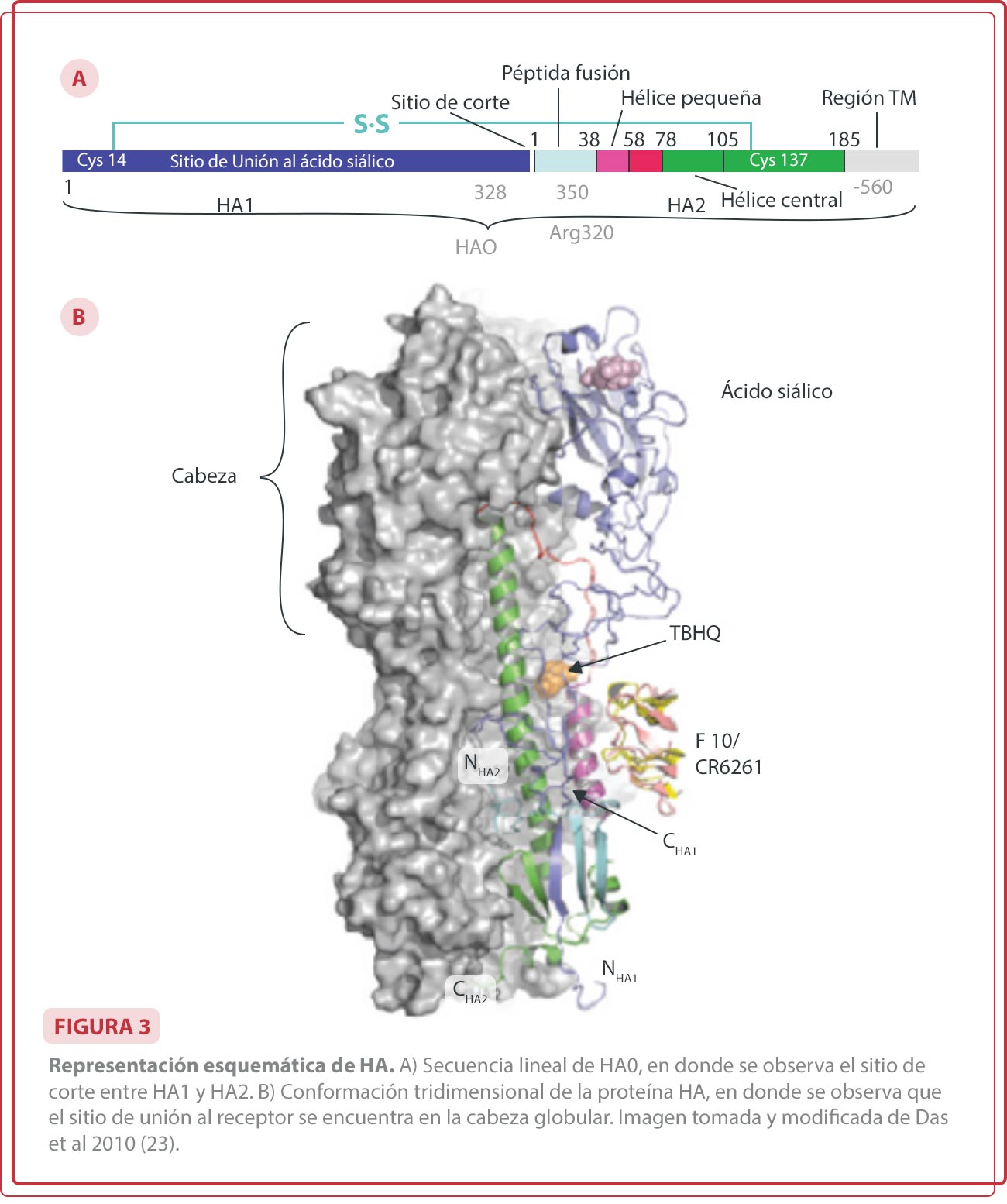
La proteína HA es codificada por el segmento 4, está compuesta por 560 aa, participa en el reconocimiento e ingreso del virus a la célula huésped. Es una glicoproteína de superficie, que constituye aproximadamente el 80% de las proteínas expresadas en la superficie viral (18). Se encuentra formando homotrímeros de forma cilíndrica (Figura 3), cada homotrímero consiste en un tallo fibroso, que en un extremo se inserta en la membrana viral, mientras que en el otro extremo se encuentra el dominio globular con tres sitios de unión para el ácido siálico (un sitio por cada monómero de HA). Los monómeros de HA son sintetizados como HA0, este precursor es inactivo, por lo que no puede infectar hasta ser cortado por enzimas proteolíticas. En los virus aviares el sitio de corte determina la patogenicidad de la cepa viral, los de baja patogenicidad tienen un sitio de corte monobásico. Los virus de baja patogenicidad son digeridos por enzimas proteolíticas tipo tripsina (19), que se encuentran en el tracto respiratorio y en el tracto gastrointestinal, y de esta manera la infección viral es limitada (20, 21). Los virus de alta patogenicidad contienen un sitio de corte polibásico, con una mayor cantidad de argininas y lisinas, lo que permite que sean digeridos por enzimas intracelulares como las proteasas ubicuas, resultando en una diseminación viral sistémica. Actualmente se han identificado 18 subtipos de HA (22).
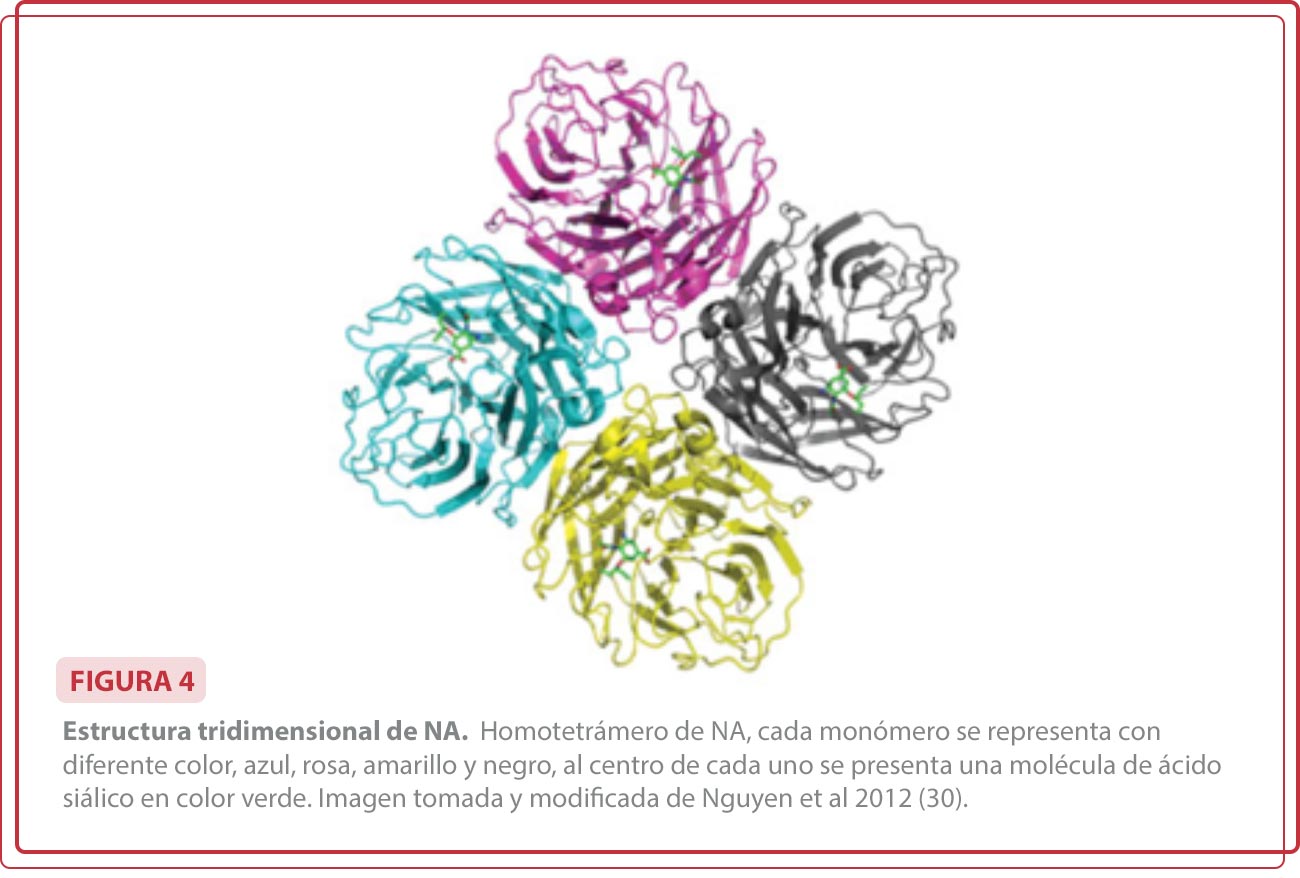
La proteína NA es codificada por el segmento 6, está compuesta por 465 aa, participa en la liberación de la progenie viral. Es una glicoproteína con actividad de sialidasa, su principal función es liberar la progenie viral, cortando las uniones entre la HA y el ácido siálico de la célula infectada (24, 25, 22). La NA se encuentra en la superficie del virión formando un homotetrámero, cada monómero está compuesto por un dominio citoplasmático, una región transmembranal, un tallo hipervariable, y una cabeza globular que tiene el dominio catalítico de esta enzima, con regiones altamente conservadas en su sitio activo (Figura 4) (26). Entre las funciones de la NA también se encuentran prevenir la agregación de los viriones liberados y romper los enlaces de ácido N-acetil-neuramínico del moco para que el virus pueda establecerse en el aparato respiratorio superior (27). Los fármacos anti influenza se enfocan en inhibir la actividad de la NA y de esta manera detener la diseminación viral y el desarrollo de la enfermedad (28). Los virus de influenza que carecen de NA tienen la capacidad de replicarse, aunque la replicación de estos virus se encuentra disminuida con respecto a los virus silvestres. La replicación viral en ausencia de NA replantea la efectividad de los antivirales que se enfocan en inhibir la actividad de esta enzima (29) Actualmente se han identificado 11 subtipos de NA (22).
La infección, replicación y diseminación de los virus de influenza, está determinada por la adecuada funcionalidad de cada una de las proteínas para las cuales codifica su genoma; las mutaciones producidas durante el proceso de replicación en cada uno de los segmentos génicos puede tener efectos en como las proteínas codificadas realizan cada una de sus funciones, por lo cual es muy importante hacer un abordaje integral del estudio de todas las proteínas virales cuando detectamos un comportamiento inusual de los virus de influenza en circulación, ya que los virus de influenza como pudimos ver son mucho más que solo HA.
Referencias
1.- Vasin, A.V., Temkina, O.A., Egorov, V.V., Klotchenko, S.A., Plotnikova, M.A., Kiselev, O.I. Molecular mechanisms enhancing the proteome of influenza A viruses: An overview of recently discovered proteins. Virus Res. 2014. 185: 53–63.
2.- Martin, M., Hans, W.D., Jindrich, C.J. Novel swine-origin influenza A virus in humans: another pandemic knocking at the door. Med Microbiol Immunol. 2009. 198: 175-183.
3.- Tonga, S., Lia, Y., Rivailler, P., Conrardya, C., Alvarez-Castillo, D.A., Chen, L.M., Recuenco, S., Ellison, J.A., Davis, C.T., York, I.A., Turmelle, S.A., Moran, D., Rogers, S., Shi, M., Tao, Y., Weil, M.R., Tang, K., Rowe, L.A., Sammons, S., Xu, X., Frace, M., Lindblade, K.A., Cox, N.J., Anderson, L.J., Rupprecht, C.E., Donis, R.O. A distinct lineage of influenza A virus from bats. PNAS. 2012. 109 (11): 4269-4274.
4.- Vega, B.R.S., Reyes-Terán, G. El virus de la influenza. Neumol Cir Torax. 2007. 66: 12-14.
5.- Bhoumik, P., Hughes, A.L. Reassortment of ancient neuraminidase and recent hemagglutinin in pandemic (H1N1) 2009 virus. Emerg Infect Dis. 2010. 16: 1-7.
6.- Wohlbold, T.J., Krammer, F. In the Shadow of Hemagglutinin: A Growing Interest in Influenza Viral Neuraminidase and Its Role as a Vaccine Antigen. Viruses. 2014. 6: 2465-2494.
7.- Boivin, S., Cusack, S., Ruigrok, R.W.H.,Hart, D. J. Influenza A Virus Polymerase: Structural Insights into Replication and Host Adaptation Mechanisms. JBC. 2010. 285 (37): 28411–28417.
8.- Medina, R.A., Garcia-Sastre, A. Influenza A viruses: new research developments. Nat
Rev Microbiol. 2011. 9: 590-603.
9.- Tauber, S., Ligertwood, Y., Quigg-Nicol, M., Dutia, B.M., Elliott, R.M. Behaviour of influenza A viruses differentially expressing segment 2 gene products in vitro and in vivo. J Gen Virol. 2012. 93 (4): 840-849.
10.- Reich, S., Guilligay, D., Pflug, A., Malet, H., Berger, I., Crépin, T., Hart, D., Lunardi, T., Nanao,M., Ruigrok, R.W.H. Cusack, S. Structural insight into cap-snatching and RNA synthesis by influenza polymerase. Nat. 2014. 516: 361-366.
11.- Oishi, K., Yamayoshi, S., Kawaoka, Y. .Mapping of a region of the PA-X protein of influenza A virus that is important for its shut-off activity. J Virol. 2015. 3: 01132-15.
12.- Khaperskyy, D.A., McCormick, C. Timing Is Everything: Coordinated Control of Host Shutoff by Influenza A Virus NS1 and PA-X Proteins. J. Virol. 2015. 89 (13): 6528-6531.
13.- Resa-Infante, P., Jorba, N., Coloma, R., Ortín, J. The influenza RNA synthesis machine advances in its structure and function. RNA Biology. 2011. 8 (2): 207-215.
14.- Portela, A., Digard, P. The influenza virus nucleoprotein: a multifunctional RNA-binding protein pivotal to virus replication. J Gen Virol. 2002. 83 (4): 723-734.
15.- Townsend, A.R.M., McMichael, A.J., Carter, N.P., Huddleston, J.A., Brownlee, G.G. Cytotoxic T cell recognition of the influenza nucleoprotein and hemagglutinin expressed in transfected mouse L cells. Cell. 1984. 39(1):13-25.
16.- Noton, S.L., Medcalf, E., Fisher, D., Mullin, A.E., Elton, D., Digard, P. Identification of the domains of the influenza A virus M1 matrix protein required for NP binding, oligomerization and incorporation into virions. J Gen Virol. 2007. 88 (8): 2280–2290.
17.- Webster, R. G., Bean, W.J., Gorman, O.T., Chambers, T.M., Kawaoka, Y. Evolution and Ecology of Influenza A Viruses. Microbiol Rev. 1992. 56 (1): 152-179.
18.- Lyndon, J., Mitnaul, Mikhail, N., Matrosovich, M.R., Castrucci, A. B., Tuzikov, N. V., Bovin, D.K., Kawaoka, Y. Balanced Hemagglutinin and Neuraminidase Activities Are Critical for Efficient Replication of Influenza A Virus. J. Virol 2000. 74 (13): 6015-6020.
19.- Skehel, J.J., Wiley, D.C. Receptor Binding And Membrane Fusion In Virus Entry: The Influenza Hemagglutinin. Annual Review of Biochemistry. 2000. (69): 531-569.
20.- Steinhauer, D.A. Role of Hemagglutinin Cleavage for the Pathogenicity of Influenza Virus. Virology. 1999. 258:1-20.
21.- Sriwilaijaroen, N., Suzuki, Y. Molecular basis of the structure and function of H1 hemagglutinin of influenza virus. Proc. Jpn. Acad. 2012 .88:226-249.
22.- Tong S, Zhu X, Li Y, Shi M, Zhang J. New World Bats Harbor Diverse Influenza A Viruses. PLoS Pathog. 2013. 9(10): e1003657.
23.- Das, K., Aramini, J.M., Ma, L., Krug, R.M., Arnold, E. Structures of influenza A proteins and insights into antiviral drug targets. Nat struct mol biol. 2010. 17: 530–538.
24.- Jain, S., Fry, M.A. Peramivir: Another Tool for Influenza Treatment?. Clin Infect Dis. 2011. 52(6):707-709.
25.- Belshe, R.B. The Origins of Pandemic Influenza-Lessons from the 1918 Virus. N Engl J Med. 2005. 353 (21): 2209-2211.
26.- Li, Q., Qi, J., Zhang, W., Vavricka, C.J., Shi, Y., Wei, J., Feng, E., Shen, J., Chen, J., Liu, D., He, J., Yan, J., Liu, H., Hiang, H., Teng, M., Li, X., Gao, G.F. The 2009 pandemic H1N1 neuraminidase N1 lacks the 150-cavity in its active site. Nature structural and molecular biology. 2010. 1-3.
27.- Banks, J., Speidel, E.S., Moore, E., Plowright, L., Piccirillo, A., Capua, I., Cordioli, P., Fioretti, A., Alexander, D.J. Changes in the hemagglutinin and neuraminidase genes prior to the emergence of highly pathogenic H7N1 avian influenza viruses in Italy. Arch virol. 2001. 146: 963-973.
28.- Moscona, A. Neuraminidase Inhibitors for Influenza. N Engl J Med. 2005. 353: 1363-73.
29.- Inoue, E., Ieko, M., Takahashi, N., Osawa, Y., Okazaki, K. An NA-deficient 2009 pandemic H1N1 influenza mutant virus can efficiently replicate in cultured cells. Arch Virol. 2013. DOI: 10.1007/s00705-013-1887-0.
30.- Nguyen, H.T., Fry, A.M., Gubareva, L.V. Neuraminidase inhibitor resistance in influenza viruses and laboratory testing methods. Antiviral Therapy. 2012. 17:159-173.
Comentarios (0)


 Español
Español  Inglés
Inglés 
