Para dejar un comentario Regístrate aquí o si ya tienes una cuenta debes Iniciar sesión

Porcinos
agosto 18, 2021
Variabilidad genética del parvovirus porcino y su implicación en la vacunación
Víctor Manuel Carrera Aguirre. Colaboradores: Ana Lucero Cebreros Cota, Juan Roberto Vázquez López, Jesús Munguía Rosas
ANTECEDENTES
El Parvovirus Porcino (PPV, recientemente llamado Ungulate Protoparvovirus 1) fue aislado por primera vez en 1965 en Munich, Alemania, por Anton Mayr y sus colaboradores, como contaminante de un cultivo de células primarias porcinas utilizado para la propagación del virus de la Peste Porcina Clásica [Mayr and Mahnel, 1964].
El Parvovirus Porcino causa trastornos reproductivos en cerdas: Mortinatos, Momificación, Muerte Embrionaria e Infertilidad, por sus siglas en ingles SMEDI (Stillbirth, Mummification, Embryonic Death and Infertility) [Mengeling et al., 2000]
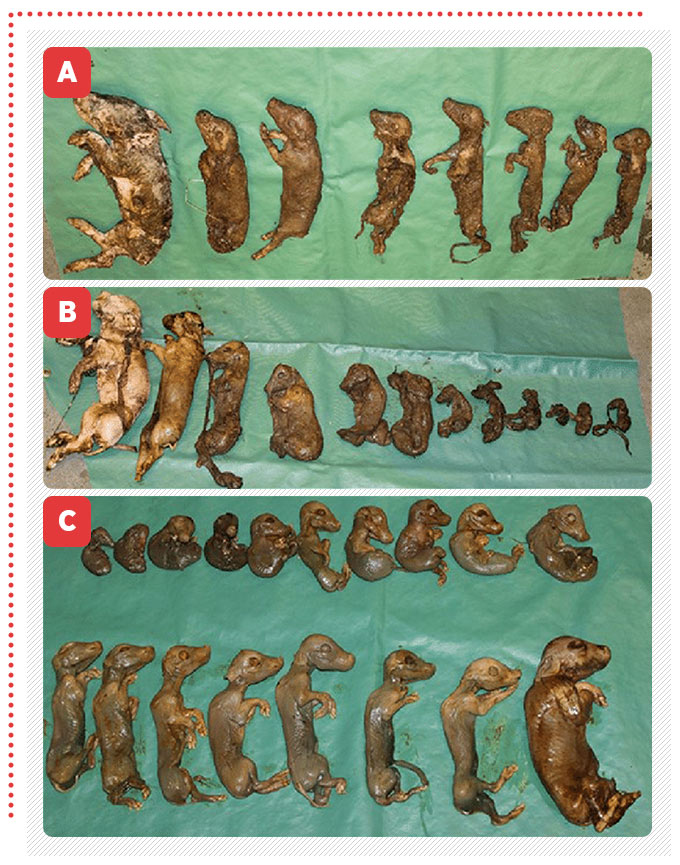 La insuficiencia reproductiva causada por parvovirus es provocada por los efectos que tiene directamente sobre los embriones y fetos. Debido a la ausencia de la respuesta inmunológica, el virus puede replicarse en el tejido uterino, por lo que, cuando ocurre la muerte de los embriones, la mayoría de las células contienen grandes cantidades de antígeno vírico intracitoplasmático [Zimmerman et al., 2012]. En la mayoría de las granjas porcinas la enfermedad es enzoótica, la mayor parte de las hembras reproductoras se infecta de manera natural y con pocas excepciones las hembras son inmunes. Una gran proporción de hembras primerizas se infectan naturalmente con PPV antes de ingresar al hato reproductor [Zimmerman et al., 2012; Xiao et al., 2013].
La insuficiencia reproductiva causada por parvovirus es provocada por los efectos que tiene directamente sobre los embriones y fetos. Debido a la ausencia de la respuesta inmunológica, el virus puede replicarse en el tejido uterino, por lo que, cuando ocurre la muerte de los embriones, la mayoría de las células contienen grandes cantidades de antígeno vírico intracitoplasmático [Zimmerman et al., 2012]. En la mayoría de las granjas porcinas la enfermedad es enzoótica, la mayor parte de las hembras reproductoras se infecta de manera natural y con pocas excepciones las hembras son inmunes. Una gran proporción de hembras primerizas se infectan naturalmente con PPV antes de ingresar al hato reproductor [Zimmerman et al., 2012; Xiao et al., 2013].
EVOLUCIÓN Y HALLAZGOS
A pesar del uso continuo de vacunas, recientemente se han descrito varias cepas nuevas y existe un motivo de preocupación porque los cambios en solo unos pocos aminoácidos en la proteína de la cápside pueden causar diferencias marcadas en la antigenicidad y virulencia.
El PPV, es un virus que se considera tiene un genoma más conservador que otros parvovirus y virus DNA, el primer análisis evolutivo se realizó en 2011 estudiando los virus que afectan a cerdos en producción intensiva [Streck et al.,2011] así como utilizando una metodología similar con los virus que afectan a los jabalíes [Cadar et al., 2012]. Se encontraron altas tasas de mutación (aproximadamente 3-5 x 10-4) en los genes VP de ambas especies hospedadoras, con una tasa de evolución más moderada para los genes NS (aproximadamente 10-5). Al aplicar un “reloj molecular” al análisis de árboles filogenéticos, se observó que las principales divergencias se introdujeron en los últimos 10 a 30 años. Esta historia evolutiva, es similar a la de los parvovirus carnívoros y humanos, sugiriendo que las altas tasas de mutación pueden ser típicas de los parvovirus porcinos.
La variabilidad genética del PPV y el descubrimiento de nuevos perfiles de cápside son temas de especial interés. Los estudios con cepas de casos clínicos en varios países, han informado nuevos perfiles y nuevos grupos con predominio de cepas de cerdos domésticos. No está claro si los nuevos patrones de aminoácidos en los aislados de PPV reflejan la verdadera evolución viral o solo la presencia de una amplia variedad de virus "antiguos" que ahora son detectables con técnicas más sensibles. Se han descrito perfiles moleculares de nuevas cápsides con distintas propiedades antigénicas, incluidos los virus utilizados en vacunas comerciales [Zeeuwet et al., 2007].
Estos hallazgos, junto con la predicción de que las principales divergencias entre los nuevos aislamientos, han llevado a la hipótesis de que la aparición de nuevos perfiles de cápside podría deberse a la adaptación viral a las vacunas más utilizadas y por lo tanto, puede representar "mutantes de escape" en una población parcialmente inmune [Streck et al., 2011; Cadar et al., 2012]
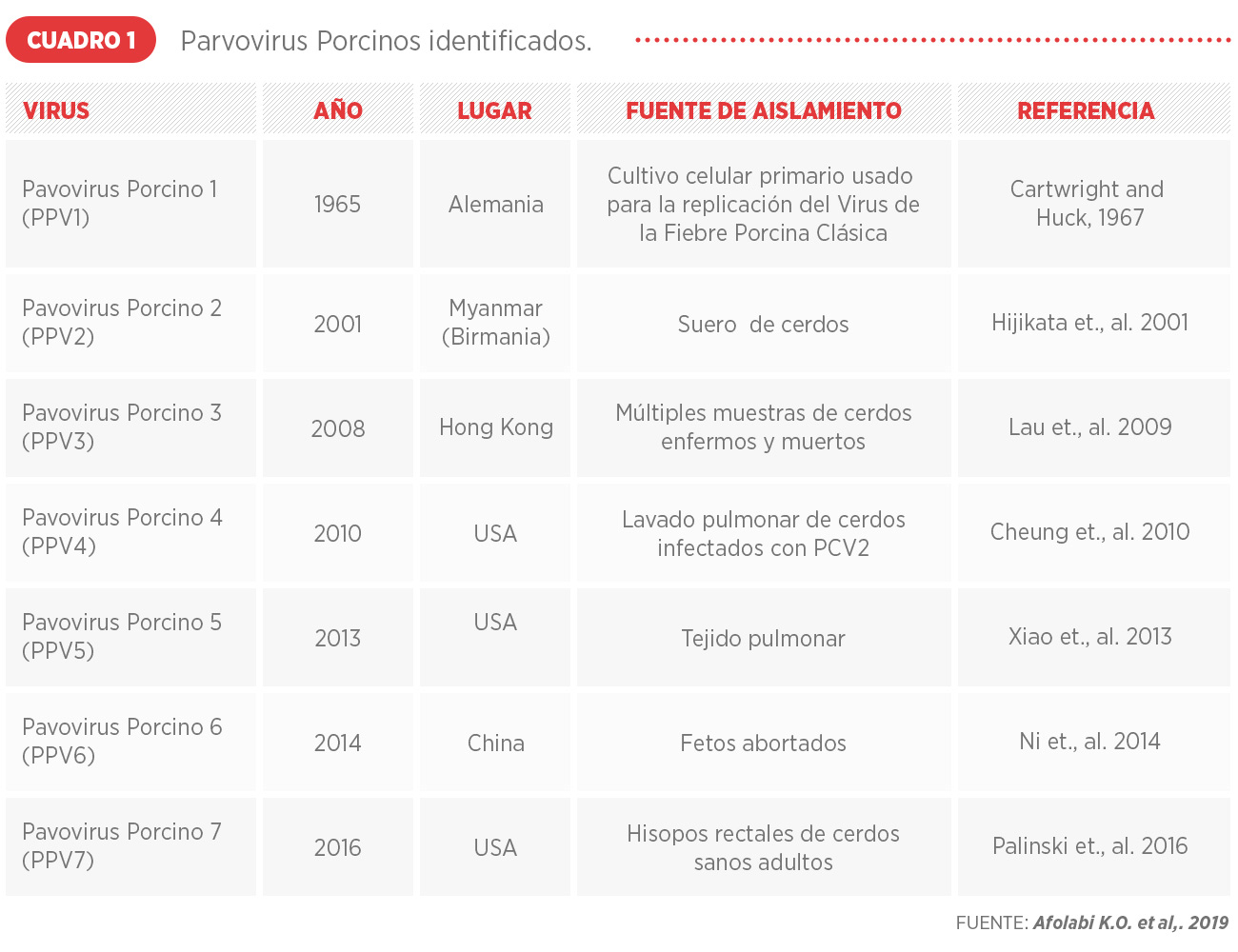
En los últimos años, con el desarrollo de nuevas técnicas moleculares, como la secuenciación de segunda generación, se han identificado por primera vez numerosas secuencias de virus. (Cuadro 1 e Imagen 1)
Hasta el momento, no se ha observado ninguna correlación con los signos clínicos para ninguno de los nuevos parvovirus y la alta prevalencia de estos virus en tejidos específicos y también en animales clínicamente sanos indica su característica comensal. El hecho de que los nuevos parvovirus porcinos se hayan encontrado en cerdos domésticos y jabalíes sugiere un flujo genético inter-especie activo [Xiao et al., 2013].
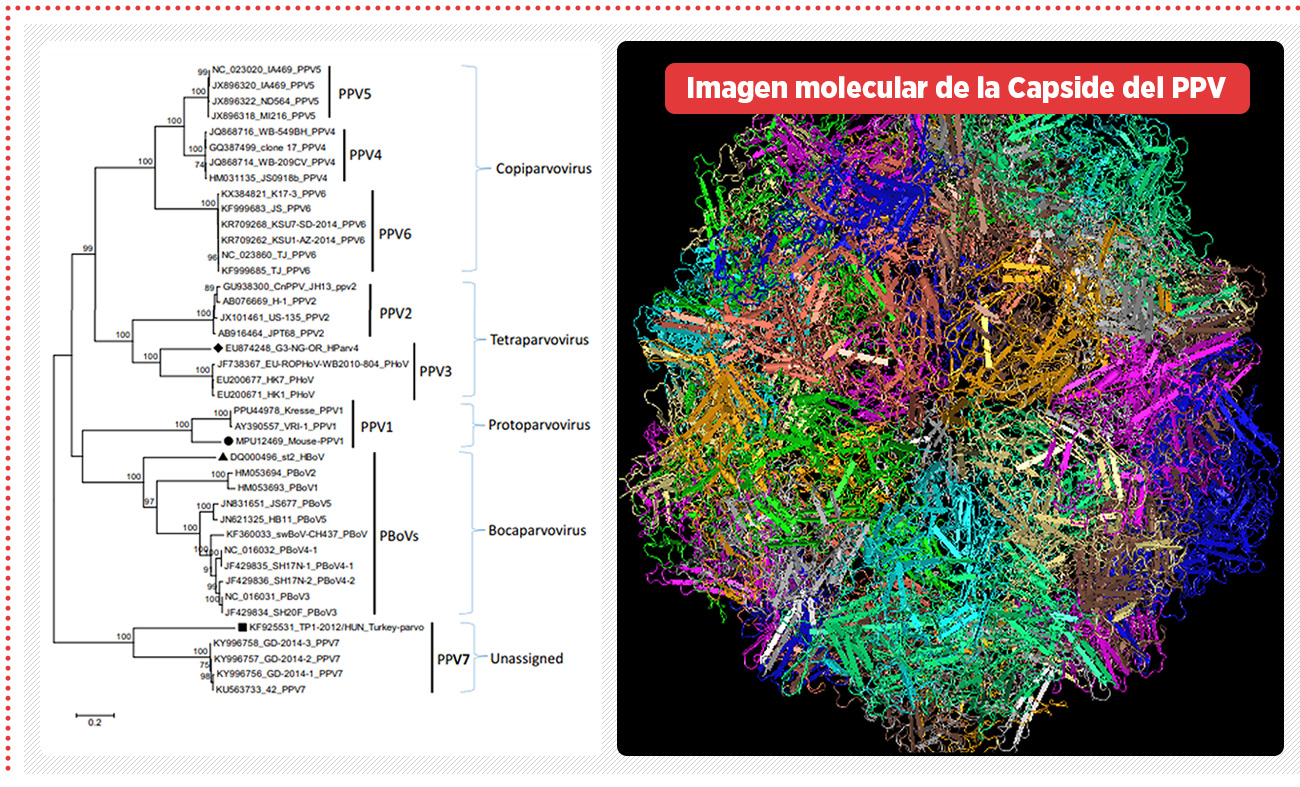
Imagen 1 Análisis filogenético basado en el gen NS1 de todos los parvovirus porcinos detectados hasta la fecha. Imagen molecular de la capside del PPV. [Tamura et., al. 2013]
VACUNACIÓN Y SEROLOGÍA
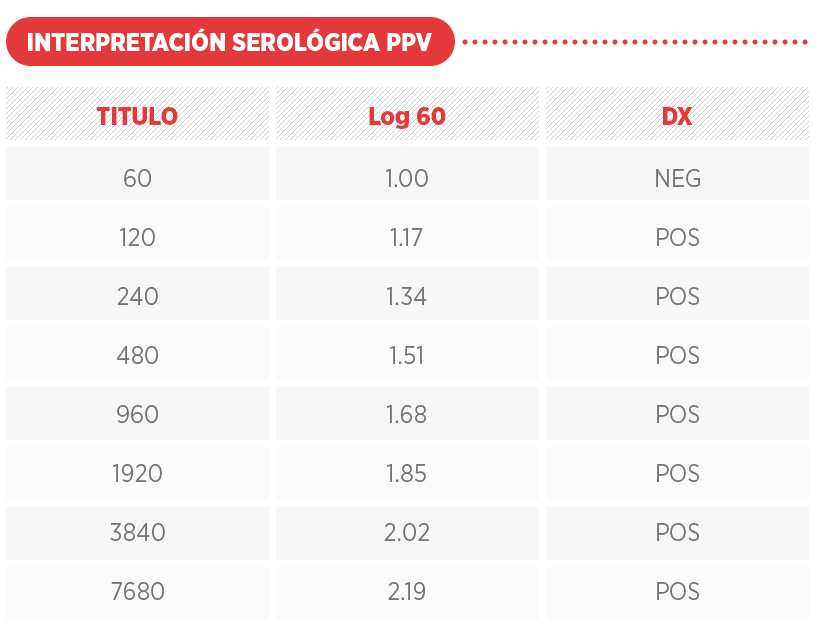
La duración de la inmunidad después de la vacunación es variable, encontrándose anticuerpos después de 2 meses post vacunación. Los títulos con vacunas comerciales son bajos (Promedio 1:480) y dispersos pero la seroconversión obtenida es considerada proyectiva.
Las vacunas actualmente disponibles en el mercado poseen cepas aisladas desde hace 40 años, basadas en la NADL-2, que son efectivas contra infecciones homólogas. Sin embargo, no previenen la infección ni eliminan el virus después de ser expuestas al desafío con la cepa antigénicamente heteróloga. Estas vacunas se elaboran con el virus inactivado (en su mayoría con cepas similares a la cepa no virulenta NADL-2) y poseen un amplio margen de seguridad; además provén protección contra el PPV aunque se administren en conjunto con otros agentes en vacunas polivalentes. El uso de estas vacunas induce buenos títulos de anticuerpos, suficientes para reducir las manifestaciones clínicas pero incapaces de prevenir la infección o diseminación [Jóźwik et al., 2009]. Las vacunas de virus vivos modificadas pueden ser una alternativa para el PPV.
 Es de suma importancia de que las hembras de reemplazo tengan un calendario adecuado de vacunación y exposición natural (feedback) para la prevención de la enfermedad antes de la entrada a su etapa reproductiva. El perfil serológico, nos proporciona información vital para conocer los problemas actuales que representan la suma de varios trastornos subclínicos que suceden en diferentes momentos de la producción. Es posible establecer seroperfiles para la vacunación o como una base a partir de la cual pueden demostrarse desviaciones del perfil normal cuando se encuentre un problema de campo o vacunación.
Es de suma importancia de que las hembras de reemplazo tengan un calendario adecuado de vacunación y exposición natural (feedback) para la prevención de la enfermedad antes de la entrada a su etapa reproductiva. El perfil serológico, nos proporciona información vital para conocer los problemas actuales que representan la suma de varios trastornos subclínicos que suceden en diferentes momentos de la producción. Es posible establecer seroperfiles para la vacunación o como una base a partir de la cual pueden demostrarse desviaciones del perfil normal cuando se encuentre un problema de campo o vacunación.
A continuación, se muestran perfiles serológicos ante desafíos de campo y vacunación con el uso de biológicos comerciales. Ver cuadro de interpretación serológica Log60.
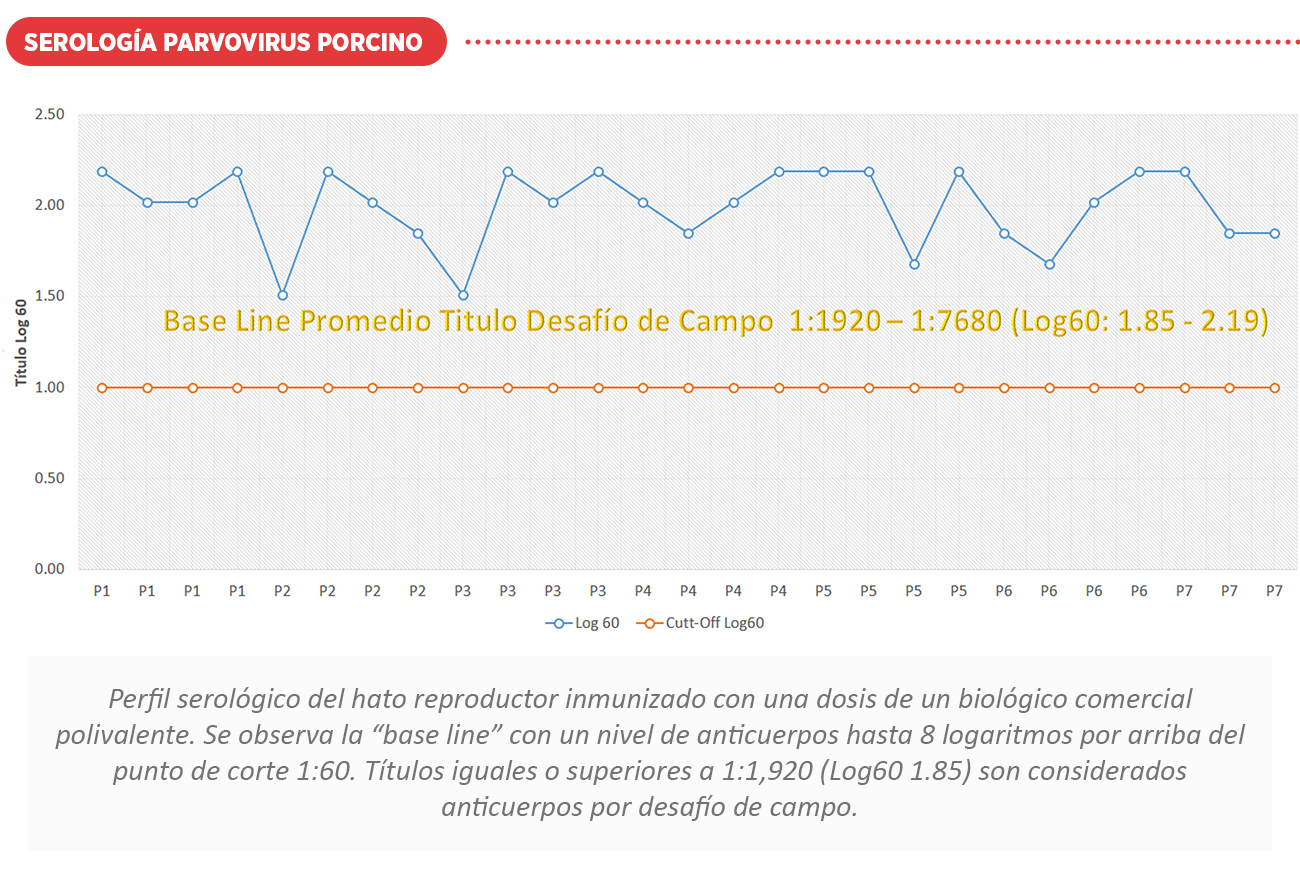
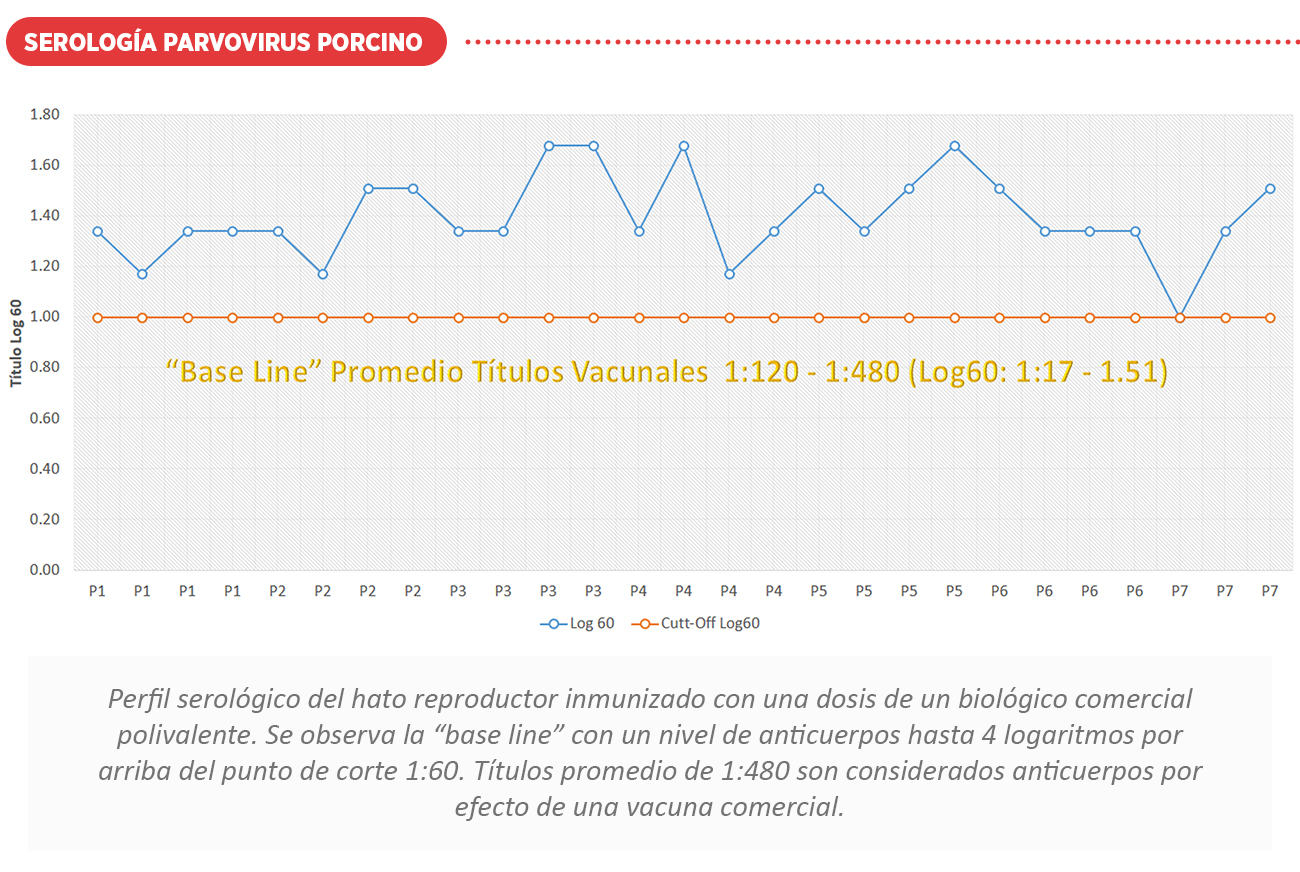
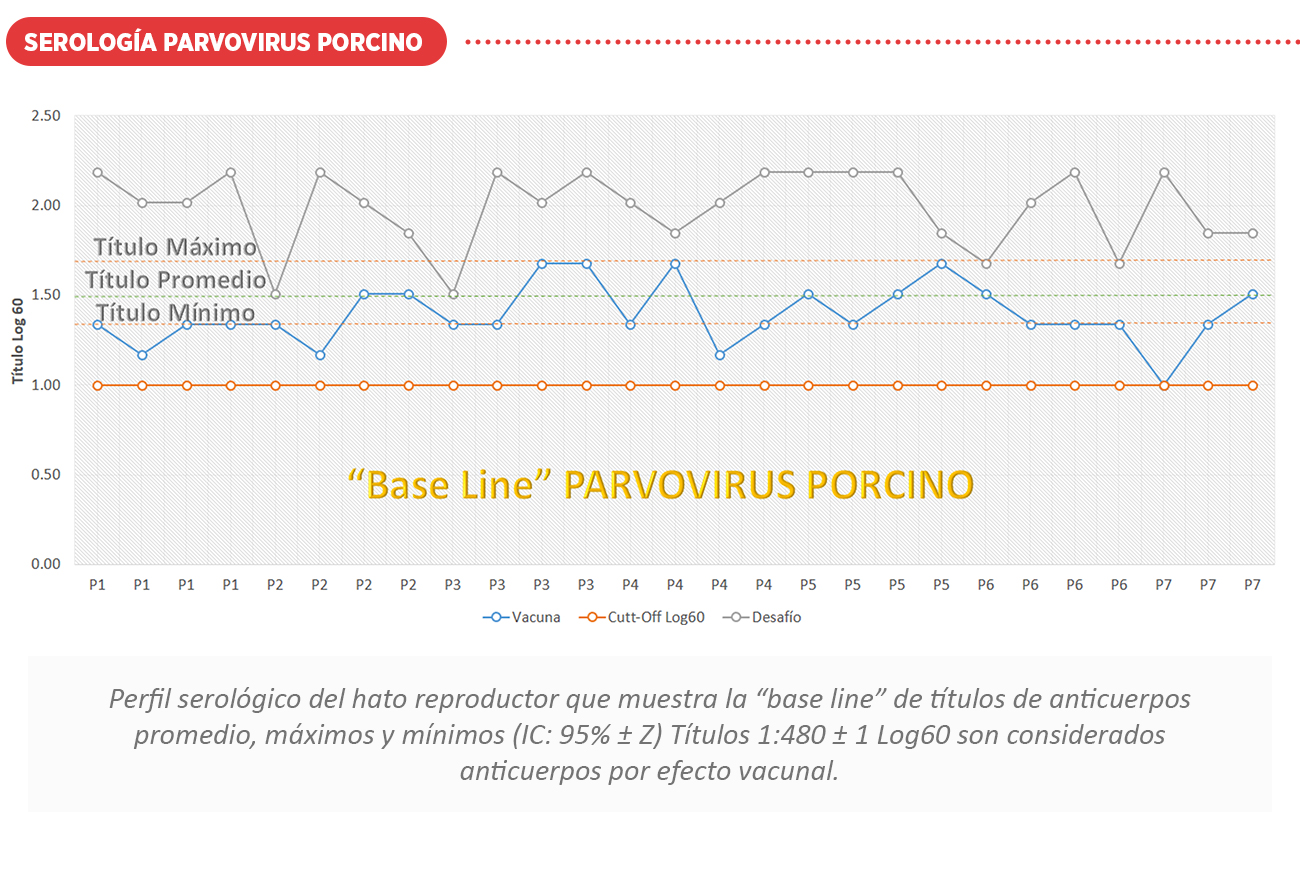
CONCLUSIONES
- El parvovirus porcino es una de las causas más importantes de falla reproductiva. En los últimos años se han observado nuevos cambios amínicos localizados en la superficie de la cápside viral creando un perfil distinto al de las cepas vacunales.
- Anteriormente, se creía que el PPV era un virus muy estable porque las vacunas desarrolladas a partir de cepas antiguas conferían inmunidad contra sus variantes, sin embargo, con el descubrimiento de nuevos perfiles de cápside esta situación ha cambiado y crea un entorno desafiante para la generación de nuevos biológicos que confieran mayor protección.
- Hoy en día, la porcicultura mundial requiere nuevas formulaciones de vacunas que induzcan una inmunidad más eficiente y que la protección tenga un alcance a todas las cepas virales circulantes, ya que las vacunas comerciales actualmente disponibles carecen de una protección completa.
REFERENCIAS BIBLIOGRÁFICAS
1) Cadar, D., Dán, Á., Tombácz, K., Lőrincz, M., Kiss, T., Becskei, Z., Spînu, M., Tuboly, T., Cságola, A., 2012. Phylogeny and evolutionary genetics of porcine parvovirus in wild boars. Infect. Genet. Evol. 12, 1163–1171.
2) Jóźwik A, Manteufel J, Selbitz HJ y Truyen U. 2009. Vaccination against porcine parvovirus protects against disease, but does not prevent infection and virus shedding after challenge infection with a heterologous virus strain. Journal of General Virology, 2437-2441
3) Kayode Olayinka Afolabia, Benson Chuks Iweriebor, Anthony Ifeanyi Okoh, Larry Chikwelu Obi. Infection, Genetics and Evolution 73 (2019) 175–183
4) Tamura, K., Stecher, G., Peterson, D., Filipski, A., Kumar, S., 2013. MEGA6: molecular evolutionary genetics analysis version 6.0. Mol. Biol. Evol. 30, 2725–2729.
5) Mayr, A., Mahnel, H., 1964. Cultivation of hog cholera virus in pig kidney cultureswith cytopathogenic effect. Zentralbl. Bakteriol. Orig. 195, 157–166.
6) Mengeling, W.L., Lager, K.M., Vorwald, A.C., 2000. The effect of porcine parvovirus and porcine reproductive and respiratory syndrome virus on porcine reproductive performance. Anim. Reprod. Sci. 60–61, 199–210.
7) Streck, A.F., Bonatto, S.L., Homeier, T., Souza, C.K., Gonçalves, K.R., Gava, D., Canal, C.W., Truyen, U., 2011. High rate of viral evolution in the capsid protein of porcine parvovirus. J. Gen. Virol. 92, 2628–2636.
8) Xiao, C.T., Giménez-Lirola, L.G., Jiang, Y.H., Halbur, P.G., Opriessnig, T., 2013. Characterization of a novel porcine parvovirus tentatively designated PPV5. PLoS One 8, 65312.
9) Zeeuw, E.J.L., Leinecker, N., Herwig, V., Selbitz, H.J., Truyen, U., 2007. Study of the virulence and cross-neutralization capability of recent porcine parvovirus field isolates and vaccine viruses in experimentally infected pregnant gilts. J. Gen. Virol. 88, 420–427.
10) Zimmerman, J. et al.(2012) DISEASES OF SWINE. WILEY-BLAC, Disease of Swine: 9th Edition. Ames, Iowa: Blackwell Publishing,. WILEY-BLAC. USA. doi: 10.1017/CBO9781107415324.004.
Comentarios (0)


 Español
Español  Inglés
Inglés 
